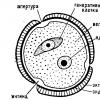
Важной инновацией молекулярной биологии стала технология биологических микрочипов. Эта технология позволяет использовать сравнительно небольшие количества исходного материала, проводить реакцию в микрообъемах, одновременно осуществлять многопараметрический анализ множества генов одного и того же объекта.
Ее чувствительность сопоставима со стандартными амплификационны- ми методами ДНК-диагностики и в некоторых случаях превосходит их .
В зависимости от природы иммобилизованных зондов различают 4 основные типа биочипов:
ДНК-чипы;
РНК-чипы;
Белковые микрочипы;
Клеточные микрочипы.
Наибольшее распространение в исследовательской и клинической практике, особенно для анализа спектра различных мутаций или
аллельных вариантов разных генов, получили ДНК-чипы. В типичном варианте они представляют собой миниатюрные гелевые пластинки с многочисленными углублениями, ячейками, содержащими набор ДНК-зондов (рис. 4.10), расположенными на стекле или мембране. За последние десять лет технология микрочипов превратилась в бурно развивающееся прикладное направление биологической науки: десятки фирм разрабатывают и предлагают биологические микрочипы, содержащие массивы от нескольких десятков до сотен тысяч и более ДНК-зондов . С помощью биочипов можно также анализировать и такие изменения ДНК, как транслокации, дупликации, протяженные делеции, а также микродупликации и микроделеции .
Технологии изготовления ДНК-чипов различаются размером наносимых фрагментов ДНК, методами иммобилизации, процедурами гибридизации и системами детекции . По конечному этапу детекции микрочипы бывают двух типов: гибридизационные и ферментативные. Гибридизационные чипы в зависимости от способа считывания сигнала подразделяются на электронные (фирма “Nanogen”) и флюоресцентные (фирмы “Affymetrix”, “Illumina”, «Биочип») и т. д. [Биочип: www.biochip. rn; Affymetrix: www.affymetrix.com; Applied Biosystems: www.europe. appliedbiosystems.com; Asper Biotech: www.asperbio.com; Illumina: www. illumina.com; Nanogen: www.nanogen.com].
При этом дискриминация мутаций может происходить как до гибридизации, то есть еще в процессе подготовки пробы (чипы фирм “Applied Biosystems”, “Affymetrix”), так и в процессе гибридизации (фирма «Биочип»). Для примера проиллюстрируем гибридизацион-
 |
Рис. 4.11. Метод анализа мутаций на основе аллель-специфичной гибридизации, разработанный в ИМБ РАН (пояснения в тексте)
ные методы анализа мутаций, используемые фирмами «Биочип» (1) и «Affymetrix» (2).
1. Предложенная и разработанная в России технология микрочипов (Институт молекулярной биологии им. В. А. Энгельгардта РАН) предполагает предварительную мультиплексную амплификацию исследуемых фрагментов ДНК с использованием флюоресцентно меченных праймеров (или дезоксинуклеотидтрифосфатов) . В результате добавления избытка одного из праймеров и проведения 2 раундов репликации достигается образование большого количества преимущественно меченого одноцепочечного продукта. Последний наносят на биочип, где он гибридизуется с иммобилизованными в геле олигонуклеотидными зондами (рис. 4.11). Если последовательность анализируемой ДНК полностью комплементарна последовательности ДНК-зонда, образуется стабильный дуплекс, который легко детектируется благодаря флюоресцентной метке. Однако, если искомого фрагмента нет или в нем присутствует некомплементарное основание, то стабильный дуплекс не возникает (сигнал флюоресценции отсутствует). Данный метод позволяет анализировать до 50 полиморфных вариантов с точностью более 98 %.
2. Для анализа генетического полиморфизма и мутаций, используя технологию “Affymetrix” , предполагается при-
менение так называемых «специальных проб». Такие пробы состоят из нескольких фрагментов: H1 и H2 - специфичных для анализируемой последовательности ДНК; P1 и P2 - являющихся универсальными праймерами и фланкирующих сайт для фермента клевазы; и tag-последовательности - специфической метки для выявления определенного SNP при гибридизации на микрочипе (рис. 4.12). Детекция мутаций происходит следующим образом: в исследуемый образец, содержащий одноцепочечную ДНК с искомой мутацией, добавляют «специальную пробу». Проба комплементарно гибридизуется с последовательностью ДНК анализируемого образца (фрагментами H1 и H2) (рис. 4.12 А). Далее добавляют термостабильную ДНК-полимеразу и четыре разных нуклеозидтрифосфата (используют, соответственно, четыре пробирки). ДНК-полимераза достраивает 3’-конец пробы H1 на одно основание, соответствующее вариабельной позиции (рис. 4.12 B). Затем проводят лигазную реакцию, при которой происходит соединение 5’-конца встроенного основания с 3’-концом пробы H2 (рис. 4.12 С). На следующей стадии (рис. 4.12 D) экзонуклеаза разрушает оставшуюся ДНК исходного образца. С целью дальнейшей амплификации кольцевую молекулу пробы разрезают клевазой (рис. 4.12 E). Амплификацию проводят с использованием универсальных праймеров P1 и P2 (рис. 4.12 F). Далее пробы гибридизуют с микрочипом. Специфичность гибридизации достигается за счет специально сконструированного tag-фрагмента, который, наряду с универсальными праймерами, является одним из ноу-хау фирмы “Asymetrix”. Таким образом, в исследуемом образце можно однозначно выявлять до 1000 и более SNPs. Точность метода при анализе тысячи SNPs составляет около 90 %.
3. Еще один из перспективных вариантов биочипов для анализа генетического полиморфизма сконструирован на основе метода SBE (single base primer extension - удлинение праймера на одно основание), позволяющего достигать высокой степени дискриминации гиб- ридизационных сигналов на аллелях «мутантного» и «дикого» типов. При подготовке проб предварительно амплифицируют исследуемый фрагмент ДНК, содержащий маркерный SNP, после чего проводят его гибридизацию на биочипе. Последовательность зонда должна быть комплементарна последовательности исследуемой ДНК, включая его последнее основание на З’-конце, после которого следует вариабельный нуклеотид. Принцип детекции заключается в следующем: после гибридизации зонда с образцом в реакцию добавляют меченные разными красителями дидезоксинуклеотиды и ДНК-полимеразу. Благодаря присутствию дидезоксинуклеотидов возможно присоединение только одного-единственного нуклеотида к 3’-концу иммобилизованного зонда. После отмывки чипа флюоресценция тестируемой пробы определяется именно этим меченым дидезоксинуклеотидом. По степени дискриминации между гомозиготными и гетерозиготными генотипами метод SBE в среднем на порядок превосходит гибридизацию с аллель- специфичными зондами . Недостатки этого метода состоят в необходимости синтеза олигонуклеотидов, иммобилизованных на чипе со свободным З’-концом, что исключает применение метода для наиболее перспективного типа чипов, создаваемых способом фотолитографии.
4. Концептуально близкой технологии SBE являются биочипы, созданные на основе метода APEX (arrayed primer extension - достройка праймеров, синтезированных на обеих цепях ДНК). Отличие состоит в том, что при анализе тестируется не одна, а сразу обе цепи ДНК и используются несколько различных иммобилизованных зондов для каждой позиции. Это дает возможность идентифицировать с высокой степенью надежности новые мутации и полиморфные сайты. Чипы на основе APEX-технологии разработаны эстонской фирмой «Asper Biotech» , показаны на рисунке 4.13.
5. Несколько вариантов гибридизационных биочипов были разработаны на базе НИИ АГ им. Д. О. Отта СЗО РАМН. С помощью одного из
|
|||||||||||||||||||||
|
|||||||||||||||||||||
|
|||||||||||||||||||||
|
|||||||||||||||||||||
|
|||||||||||||||||||||
|
|||||||||||||||||||||


них - «фармакогенетического биочипа» - можно изучать наследственную предрасположенность к привычной невынашиваемости беременности и к лейкозам у детей . Биочип позволяет проводить анализ 13 аллельных полиморфных сайтов 7 генов системы детоксикации: CYP1A1 (4887С>А,4889A>G и 6235T>C), CYP2D6 (1934G>A и 2637delA), GSTM1 (делеция), GSTT1 (делеция), NAT2 (481T>C,590A>G и 857A>G), CYP2C9 (430С>Г и 1075C>T) и CYP2C19 (681G>A) и одного (677C>T) гена метилентетрагидрофолиевой кислоты - MTHFR. На рисунке 4.14 приведены результаты биочипового анализа с применением специального программного обеспечения. «Фармаген-биочип» уже прошел клинические испытания и внедрен в практику лабораторной диагностики нескольких медицинских центров РФ. На стадии клинических испытаний находятся биочипы для тестирования наследственной предрасположенности к тромбофилии («ТРОМБО-биочип») и к сердечно-сосудистым заболеваниям («Кардиобиочип») . Ведется работа по созданию биочипов по тестированию основных мутаций в гене СFTR при муковисцидозе, а также наследственной предрасположенности к бронхиальной астме и остеопорозу.
6. Особого внимания заслуживают биочипы, позволяющие осуществлять общегеномный скрининг ассоциаций (Genome Wide Association Studies - GWAS) ,
![]() Nsp I Nsp I Nsp I
Nsp I Nsp I Nsp I

RE Digestion
в результате которого становится возможной идентификация всех генов-маркеров, геномных локусов и отдельных маркерных SNP, ассоциированных с различными МФЗ (см. гл. 2, 3, 9). С этой целью исследуемый образец ДНК подвергают гидролизу определенными эндонуклеазами, образовавшиеся фрагменты лигируют (сшивают) с адаптерными последовательностями ДНК и амплифицируют с одним праймером, специфичным исследуемому фрагменту генома. Полученные фрагменты ПЦР метят флюоресцентными красителями и гибридизуют с наборами ДНК-зондов, находящихся на биочипе (рис. 4.15). По результатам анализа гибридизационной картины биочипа с помощью специальной компьютерной программы судят о наличии в исследованном образце того или иного набора аллельных вариантов однонуклеотидных замен - SNP (рис. 4.16). Сопоставление частот соответствующих аллелей у больных и здоровых индивидов позволяет идентифицировать все SNP и, соответственно, все гены и ДНК-ло- кусы, ассоциированные с конкретной болезнью, то есть выяснить специфический генетический профиль МФЗ. Метод позволяет в одном анализе исследовать до нескольких десятков и сотен тысяч маркеров, однако его точность не превышает 90 %. Поэтому на современном эта-

пе данный метод следует рассматривать скорее как поисковый, но не диагностический. В настоящее время метод с успехом применяется для полногеномного скрининга ассоциаций различных МФЗ, включая диабет тип 1 и тип 2, коронарную болезнь, бронхиальную астму, болезнь Крона, маниакально-депрессивный психоз и др. (см. разделы 1, 6.1, 6.3, 9).
7. Существенным продвижением в повышении эффективности, точности и стоимости тестирования мутаций явились разработки последних двух лет, направленные на совмещение несомненных преимуществ метода ПЦР в реальном времени с принципами биочиповой диагностики (рис. 4.17). Так, с помощью биочипа фирмы “Fluidigm” можно одновременно анализировать мутации или тестировать SNP в 9216 локусах. Метод имеет большую специфичность и точность, чем метод полногеномного скрининга GWAS (см. раздел 4.6.). Себестоимость исследования одной мутации/поли- морфизма не превышает 1 рубля.
Use your existing TaqMan assays: 99% assay conversion rate from 384-well system
Образец добавляют к микросферам, аналит связывается с микросферами
Рис. 4.19. Гибридизация на микросферах (www.perkinelmer.com) (пояснения в тексте)
ДНК-микрочип (англ. DNA microarray) - это сложная технология, используемая в молекулярной биологии и медицине. ДНК-микрочип представляет собой небольшую поверхность, на которую с большой плотностью в определённом порядке нанесены фрагменты одноцепочечной синтетической ДНК с известной последовательностью. Эти фрагменты выступают в роли зондов, с которыми гибридизуются (образуют двуцепочечные молекулы) комплементарные им цепи ДНК из исследуемого образца, обычно меченные флуоресцентным красителем. Чем больше в образце молекул ДНК с определенной последовательностью, тем большее их количество свяжется с комплементарным зондом, и тем сильнее будет оптический сигнал в точке микрочипа, куда был «посажен» соответствующий зонд. После гибридизации поверхность микрочипа сканируется, и в результате каждой последовательности ДНК ставится в соответствие тот или иной уровень сигнала, пропорциональный числу молекул ДНК с данной последовательностью, присутствующих в смеси.
В обычном ДНК микрочипе (н-р, производства Affymetrix) зонды прикрепляются к твердой поверхности - стеклянному или силиконовому чипу. Другие платформы, например, выпускаемые Illumina, используют микроскопические шарики вместо больших твердых поверхностей. Технология ДНК-микрочипов находит самые разнообразные применения в современной биологии и медицине для анализа сложных смесей ДНК - например, совокупности всех транскриптов (матричных РНК) в клетке. ДНК микрочипы используют для анализа изменения экспрессии генов , выявления однонуклеотидных полиморфизмов , генотипирования или повторного секвенирования мутантных геномов . Микрочипы отличаются по конструкции, особенностям работы, точности, эффективности и стоимости.
Пример использования ДНК-микрочипа
Ниже приводится пример эксперимента с использованием ДНК-микрочипа.

- Выделяются или выращиваются биологические образцы, которые необходимо сравнить. Они могут соответствовать одним и тем же индивидуумам до и после какого-либо лечения (случай парных сравнений), либо различным группам индивидуумов, например, больным и здоровым, и т. д.
- Из образца выделяется очищенная нуклеиновая кислота, являющаяся объектом исследования: это может быть РНК в исследовании профиля экспрессии генов , ДНК при изучении сравнительной геномной гибридизации и т.д. Данный пример соответствует первому случаю.
- Проверяется качество и количество полученной нуклеиновой кислоты. Если требования соблюдены, эксперимент может быть продолжен.
- На основе имеющихся образцов РНК в процессе обратной транскрипции синтезируются последовательности комплементарных ДНК (кДНК, англ. cDNA).
- В процессе амплификации (синтеза дополнительных копий ДНК) количество последовательностей кДНК в образцах многократно увеличивается.
- К концам последовательностей кДНК присоединяются флуоресцентные или радиоактивные метки.
- Полученные образцы в смеси с необходимыми химическими веществами через микроскопическое отверстие наносятся на ДНК-микрочипы и начинается процесс гибридизации, в ходе которого одна из цепей кДНК присоединяется к комплементарной ей цепи, имеющейся на микрочипе.
- После окончания процесса гибридизации чипы промываются для удаления остатков материала.
- Полученные микрочипы сканируются при помощи лазера. На выходе получается одно- или двухцветные изображения (в зависимости от количества использованных красителей).
- На каждое изображение накладывается сетка, так, что каждой её ячейке соответствует участок чипа с пробами одного типа. Интенсивности свечения проб в ячейке сетки ставится в соответствие некоторое число, которое, в самом первом приближении, может служить мерой количества присутствовавших последовательностей РНК в соответствующем образце.
Дальнейшая обработка результатов требует многоэтапного привлечения сложного статистического аппарата.
Предобработка данных эксперимента
Корреляция между интенсивностями двух проб одного ДНК-микрочипа, представляющих один и тот же ген, обычно превышает 95%. Часто этот факт интерпретируют как подтверждение хорошей воспроизводимости экспериментов с чипами. Однако, если один и тот же биологические материал разделить на две части и сделать с ними разные микрочипы, корреляция между полученными интенсивностями, скорее всего, будет составлять от 60 до 80%. Корреляция на чипах с образцами, взятыми у мышей из одного помёта, может опускаться до 30%. Если эксперименты проводятся в разных лабораториях, корреляция между их результатами может быть ещё ниже .
Такая низкая воспроизводимость интенсивностей связана с совокупным воздействием большого количества источников вариации. Их можно разделить на три большие группы. Биологическая вариация включает неотъемлемые особенности организмов. Техническая вариация появляется на этапе выделения образцов, их окрашивания и гибридизации. Погрешность измерения связана со сканированием готовых массивов, на результаты которого может повлиять, например, пыль внутри сканера.
Нейтрализация эффектов технической вариации и ошибки измерения производится на этапе предобработки ДНК-микрочипов.
Фоновая поправка
Необходимость фоновой поправки связана с наличием таких мешающих факторов, как шум оптической системы распознавания (данные интенсивности, полученные при сканировании, не равны "настоящим" интенсивностям проб) и неспецифическая гибридизация (присоединение нуклеотидных последовательностей к зондам чужих проб).
Нормализация
Нормализация данных позволяет сделать несколько рассматриваемых в эксперименте чипов пригодными к сравнению между собой. Основная цель анализа на этом этапе - исключить влияние систематических небиологических различий между микрочипами. Источников таких различий множество: вариации эффективности обратной транскрипции, маркировки красителями, гибридизации, физические различия между чипами, небольшие различия в концентрации реагентов, вариация лабораторных условий.
Показано, что выбор метода нормализации оказывает существенное влияние на результат анализа .
Суммаризация
Обобщение значений уровня экспрессии по всем пробам, соответствующим одинаковым последовательностям
Контроль качества
Обработка выбросов
Основной этап статистической обработки
Ссылки
- DNA microarray
- DNA microarray experiment - статья из английской Википедии
- DNA Microarray Virtual Lab - пошаговый интерактивный пример эксперимента с двукрасочным ДНК-микрочипом
- Ten Pitfalls of Microarray Analysis - распространённые ошибки анализа ДНК-микрочипов
Юная калифорнийская компания Affymetrix (начавшая свою работу в 1993 году) - один из фаворитов рынка устройств для генетических исследовательских работ.
Компания известна своим революционным соединением технологий полупроводниковой, так сказать, «микросхемной», индустрии и биохимических тестов.
ДНК-чипы от Affymetrix обширно употребляются в различных лабораториях, занятых генетическим анализом и генной инженерией.
Но обыденным людям куда увлекательнее другой продукт компании. Это прибор, схожий на микросхему, позволяющий идентифицировать 10-ки ДНК от разных животных в образчике людской еды.
bioMerieux FoodExpert-ID практически - разновидность так именуемого GeneChip.
Прибор может идентифицировать био следы в еде от 12 разновидностей млекопитающих, 5 видов домашней птицы и 16 разновидностей рыбы.
Таким макаром, он позволяет выяснить, вправду ли гусиный паштет, вызывающий у покупателя подозрения, содержит гусиную печень, а не что-то ещё.
ДНК-чип создаётся по технологиям, схожим с компьютерными, но это не электрический, а био объект (иллюстрация с веб-сайта affymetrix.com).
А, например, мусульмане могут проверить - не положили ли нерадивые изготовители свинину в «говяжьи» котлеты.
Всё это, правда, работает, только с привлечением дополнительных лабораторных способностей, так что использовать чип в «нагом» виде, на коленке, обычному потребителю не получится.
Чтоб осознать, как работает FoodExpert-ID, необходимо вспомнить самую малость из генетики: двойные спирали ДНК, составляющие их молекулы-основания - аденин, гуанин, тимин и цитозин, также то, что они могут соединяться только попарно, как будто ключи и замки.
ДНК-чип содержит мириады и мириады «располовиненных» фрагментов ДНК-кода.
Кусок поверхности чипа с молекулами-ключами (иллюстрация с веб-сайта affymetrix.com).
Поверхность чипа размером с ноготь разбита на 97 тыщ квадратиков, нареченных «особенностями».
Любая «особенность» поперечником примерно 26 микронов содержит только один ДНК-код. Поточнее много-много схожих молекул.
И они все совершенно точно относятся к одному из 33 животных.
Длина каждого куска - 17 оснований. Этого довольно для надёжной идентификации, как довольно 17 взятых попорядку в любом месте нот, чтоб найти какую-нибудь мелодию из имеющейся базы данных.
Целую россыпь разбитых кусочков ДНК экспериментаторы выделяют из эталона еды. Чего там только нет. А чего?
«Некорректные» куски генетических кодов смываются, а совпадающие - закрепляются на чипе. Красноватые шарики - флуоресцентные молекулы (иллюстрация с веб-сайта affymetrix.com).
Добавим к молекулам, составляющим генетический код, молекулы флуоресцирующего вещества. Нанесём эту смесь на поверхность FoodExpert-ID. Осталось сделать малость.
Все совпадающие куски кода объединятся со своими «родными» последовательностями в той либо другой «особенности».
Сейчас чип можно помыть водой - всё избыточное уйдёт. Чип помещают под луч лазера, и квадратики, содержащие отловленный материал будут ярко сиять. Осталось только свериться с картой чипа, чтоб выяснить - какие ДНК определены.
А по интенсивности свечения можно сделать косвенный вывод и о пропорциях свинины и говядины в нашей гипотетичной котлете.
Как лицезреем, внедрение чипа сравнимо нетрудно, и позволяет заниматься генетическим анализом лабораториям, имеющим очень обычный набор оборудования.
Но как же хитроумно создание чипа. Чтоб создавать такие биохимические шедевры автоматизировано и в массовом порядке, Affymetrix соединила принципы фотолитографии и комбинаторной химии.
Цветные квадратики - «особенности», отвечающие за идентификацию того либо другого ДНК-кода (иллюстрация с веб-сайта affymetrix.com).
Начальный продукт - кварцевая пластинка - покрывается особым реактивом, силаном, который крепко соединяется с кварцем и сформировывает строго периодичную молекулярную матрицу (с равномерной поверхностной плотностью), готовую принять нуклеотиды.
В цепочках грядущего кода основания идут вертикально ввысь, а наносят их сразу на всю поверхность, слой за слоем.
Очевидно, всякий раз на чип подают определённое вещество, и чтоб оно закрепилось исключительно в определённых «особенностях», тех микронных квадратиках, употребляют маски, подобные тем, что необходимы для производства микросхем.
Снимок прореагировавшего чипа с огромным повышением. Белоснежные, красноватые, жёлтые квадратики - участки с высочайшей концентрацией флуоресцентного вещества. Зелёные, голубые, чёрные - соответственно, со всё более и поболее с низкой (иллюстрация с веб-сайта affymetrix.com).
С основой чипа всякий раз сцепляются только те основания, что освещаются через отверстия в маске ультрафиолетом.
В этом процессе поочередного синтеза главное - всякий раз накладывать новейшую маску с микронной точностью, по другому все генетические коды на пластинке перемешаются.
Так, шаг за шагом (в пищевом чипе их 17, в других моделях компании - до 24) формируются вертикальные столбики нуклеотидных цепей, которые и делают ключи-анализаторы генов.
Эта разработка служит, естественно, не только лишь для таких смешных (на 1-ый, может быть, взор) областей внедрения, как выявление мяса поросёнка в гусином паштете, да и для полностью серьёзных исследований.
Ведь на поверхность чипа, на теоретическом уровне, можно нанести куски каких угодно генетических кодов.
Работа Affymetrix - избыточное подтверждение, что самые достойные внимания и многообещающие открытия происходят на соединениях наук и дисциплин.
Похоже на био обилие в природе, получаемое смешением генов. Не так ли?
Производственные технологии и дизайн в наибольшей степени определяют достоинства и недостатки биологических микрочипов, области их применения, ценовые характеристики и общую доступность.
Существуют два принципиально разных подхода к производству ДНК–чипов: синтез ДНК заданной последовательности непосредственно на матрице и иммобилизация на подложке заранее синтезированных олигонуклеотидов химическим путем. Технология синтеза олигонуклеотидов на подложке фотолитографическим путем запатентована и применяется компанией Affymetrix, мировым лидером в области производства ДНК – чипов, контролирующим до 70% их мирового рынка. В основе технологии лежит применение фотолабильной защитной группы для мономерных звеньев ДНК, которая удаляется с концевого остатка синтезируемого на подложке олигонуклеотида при облучении УФ – светом. Достоинством такой технологии является возможность получения чипов с очень высокой плотностью нанесения – до 100 000 точек на 1 см2. Очевидный недостаток метода – сложность и дороговизна процесса.
Технологии химической иммобилизации фрагментов ДНК на твердых подложках начали разрабатываться около 30 лет назад и в настоящий момент продолжают совершенствоваться. Общий принцип иммобилизации биологических молекул – формирование на поверхности подложки и на конце пришиваемого олигонуклеотида пары химических групп, обеспечивающей образование между ними ковалентной связи. Существует огромное количество таких способов, большинство из которых основано на взаимодействии нуклеофильной группы (например, аминогруппы), находящейся на поверхности подложки или привязанной к молекуле олигонуклеотида, с электрофильным агентом, в роли которого могут выступать тем или иным путем активированные карбоновые кислоты, моноэфиры фосфорной кислоты и т.д.
Оценка экспрессии генов с помощью ДНК микрочипов (на примере Affymetrix GeneChip )
Наиболее часто ДНК-микрочипы применяются для оценки экспрессии генов. Наиболее популярная платформа для решения этого класса задач - микрочипы Affymetrix GeneChip, использующие короткие последовательности олигонуклеотидов для выявления генов, содержащихся в образце РНК. Присутствие в образце каждого гена фиксируется при помощи совокупности зондов длиной в 25 нуклеотидов каждый. Для улучшения качества эксперимента на чипе размещается несколько копий зондов на каждую рассматриваемую последовательность.
Микрочипы Affymetrix обычно используют от 11 до 20 пар проб на каждый изучаемый ген. Одна компонента таких пар, называемая perfect match probe (PM), в точности комплементарна последовательности соответствующего гена - подразумевается, что именно его РНК будет присоединяться к PM-зонду. Такое присоединение называется специфической гибридизацией. Тем не менее, к зондам могут присоединяться нуклеотидные последовательности и других генов (неспецифическая гибридизация). Для оценки воздействия неспецифической гибридизации используется другие компоненты пары - зонды, называемые mismatch probe (MM). Последовательность нуклеотидов в них совпадает с последовательностью в соответствующих PM-пробах с заменой центрального (тринадцатого) нуклеотида на комплементарный. Соотношение интенсивности свечения PM- и MM-проб изначально использовалось для нейтрализации эффекта неспецифической гибридизации, однако более поздние исследования поставили под сомнение правильность подобного подхода.
Полногеномное генотипирование полиморфных локусов с помощью микрочипов высокой плотности (на примере Illumina Human610-Quad BeadChip )
К настоящему времени ведущими производителями (Illumina, Affymetrix, Sequenom и др.) разработаны платформы с микрочипами высокой плотности для генотипирования и анализа экспрессии генов (рис. 30)
Illumina Human610-Quad BeadChip включает более 600 тысяч однонуклеотидных полиморфизмов и маркеров вариации по числу копий генов (CNV). Каждый ОНП (SNP) для биочипа отобран на основании точной о нем информации для повышения эффективности определения ассоциации с заболеванием. В геноме человека более 10 миллионов ОНП и изучение каждого полиморфного локуса в геноме экономически нецелесообразно. Компанией Illumina разработан уникальный рациональный подход к выбору ОНП, который обеспечивает высокое качество данных генотипирования и охват всех необходимых локусов для анализа предрасположенности к заболеванию.
Рис.30. Чиповые технологии для высокопроизводительного генотипирования
В состав биочипов Illumina включен набор ОНП, называемых маркерными или таговыми (tag SNP), которые могут быть использованы в качестве прокси-маркеров (маркеров-представителей) для всех распространенных ОНП (с частотой редкого аллеля ≥ 5%) в геноме. Выбор маркерных ОНП основывается на величине неравновесия по сцеплению (r 2) между близкорасположенными полиморфными локусами. Высокий уровень r 2 между двумя ОНП, указывающий на высокую корреляцию, делает эти ОНП хорошими прокси-маркерами. При максимальном r 2 , равном 1, два ОНП находятся в полном неравновесии по сцеплению и могут служить как абсолютные прокси-маркеры, т.е. нужно генотипировать один ОНП, чтобы узнать генотип другого (The Power of Intelligent SNP Selection (www.illumina.com)). Использование маркерных ОНП дает возможность получить максимальное количество информации, высокий уровень покрытия генома и охват генов, а также уменьшить размер исследуемой выборки без снижения эффективности определения генетической ассоциации. Геномное покрытие определяется как количество ОНП, которые находятся в неравновесии по сцеплению с референсным набором локусов. В качестве референсного набора специалисты компании Illumina используют все ОНП, прогенотипированные в проекте HapMap. По данным компании Illumina, биочип Human610-Quad BeadChip обеспечивает покрытие 89% генома в европейских популяциях (CEU, HapMap), 86% генома - в азиатских (CHB+JPT, HapMap) и 58% генома – в африканских (YRI, HapMap) (при r 2 > 0.8). Среднее расстояние между маркерами на этом биочипе составляет 4,7 т.п.н., медиана – 2,7 т.п.н.
Технология Illumina’s BeadArray основана на использовании 3-микронных кремниевых микросфер (шариков – beads), которые сами собираются в микролунки или на пучках оптических волокон или на плоских кремниевых пластинах. Каждая микросфера покрыта тысячами копий специфических олигонуклеотидов, содержащих локус-специфические и адресные последовательности, по последним из которых определяется, какой шарик в какую микроячейку встроился.
На первом этапе генотипирования проводится полногеномная амплификация образцов ДНК, после которой ДНК фрагментируется (рис.31). На следующем этапе немеченые фрагменты образцов ДНК гибридизуются с соответствующими 50-нуклеотидными зондами, фиксированными на микросферах чипа. После этого для точной идентификации аллеля к тестируемому нуклеотиду с помощью фермента ДНК-полимеразы присоединяется меченый комплементарный нуклеотид. Двухэтапная детекция аллелей каждого маркера – гибридизация с 50-нуклеотидными зондами и последующая ферментативная детекция нуклеотида обеспечивают высокую селективность и специфичность идентификации аллелей (Infinium® HD DNA Analysis BeadChips (www.illumina.com)).


Рис. 31. Схема протокола генотипирования с помощью биочипов Illumina Human610 quad BeadChip (Infinium® HD DNA Analysis BeadChips (www.illumina.com).
Таким образом, в представленном учебном пособии рассмотрены основные молекулярно-генетические методы изучения наследственных болезней человека. В одной книге невозможно описать все существующие на сегодняшний день методы исследования или представить молекулярно-диагностические протоколы для каждой конкретной формы наследственной патологии. Настоящее учебное пособие дает общие представления о методологии и стратегии проведения молекулярно-генетической диагностики, раскрывает методы, наиболее часто используемые в практике современного исследователя. Предлагаемые протоколы проведения исследований, подробное описание различных методик с иллюстрациами, помогут студентам медико-биологического профиля подготовки, специализирующихся по генетике, успешно освоить молекулярно-генетические методы анализа генома человека.
СПИСОК ИСПОЛЬЗОВАННОЙ ЛИТЕРАТУРЫ
1. Бочков Н.П. Клиническая генетика. М. 2011. – 592 с.
2. Глик Б., Пастернак Дж. Молекулярная биотехнология. Принципы и применение. - Москва: Мир, 2002. - 589 с.
3. Горбунова В.Н., Баранов В.С. Введение в молекулярную диагностику и генотерапию наследственных заболеваний. Санкт-Петербург. «Специальная Литература». 1997. – 287 с.
4. Дейвис К. Анализ генома. Методы. М. 1990. – 246 с.
5. Епринцев А.Т., Попов В.Н., Федорин Д.Н. Идентификация и исследование экспрессии генов. // Учебно-методическое пособие для ВУЗов. – Воронеж. – 2008. – 63 с.
6. Жимулев И.Ф. Общая и молекулярная генетика. Н. 2003. – 479 с.
7. Инге-Вечтомов С.Г. Генетика с основами селекции. М. - 2010. – 718 с.
8. Кузьмина Н.А. Основы биотехнологии, 2005 // http://www.biotechnolog.ru/
9. Кулмамабетова Г. Пробоподготовка. Методы выделения ДНК/РНК // Современные проблемы биологии, ЕНУ, Астана, 2012, Лекция 9.
10. Меньшикова В.В. Клинико-лабораторные аналитические технологии и оборудование. М. 2007. – 240 с.
11. Молекулярная биотехнология. Принципы и применение / под ред. Н.К. Янковского. – М. : Мир, 2002. – 588 с
12. Молекулярная клиническая диагностика. Методы / под ред. С.Херрингтона, Дж.Макги. – М.: Мир, 1999. – 558с.
13. Мутовин Г.Р. Клиническая генетика. Геномика и протеомика неследственной патологии: учебное пособие. М. 2010. – 832 с.
14. Мухачева Т.А., Ковалев С.Ю. Прикладная биоинформатика. Спецкурс. УрФУ, г. Екатеринбург. 2012.
15. Патрушев Л.И. Экспрессия генов. М. Наука. 2000. – 830 c.
16. Поляничко А. М. Электрофорез в ПААГ. Методическое пособие. Санкт-Петербург. 2007.
17. Притчард Д.Д. – Наглядная медицинская генетика. – пер. с англ. под ред. Н.П. Бочкова. – М.: ГЭОТАР-Медиа. – 2009. – 200с.
18. Пузырев В.П., Степанов В.А. Патологическая анатомия генома человека. Н. – 1997. – 224 с.
19. ПЦР в реальном времени / под ред. Д.В. Ребрикова. - М.: Бином., 2009. – 223с.
20. Северин Е.С. Биохимия: Учебник. М. 2004. – 784 с.
21. Сукачев М. Современные методы полногеномного секвенирования (расшифровки) ДНК в диагностике и лечении заболеваний // http://innoros.ru/publications/articles/13/
22. Телков М. PCR реального времени: методические основы, оптимизация, применение. Bio-Red Laboratories.
23. Теоретические основы полимеразной цепной реакции. НПО «ДНК-Технология» - Москва, 1998 // www.dna-technology.ru
24. Шатц В.Д., Сахартова О.В. Высоко-эффективная жидкостная хроматография. Основы теории. Методолгия. Применение в лекарственной химии.
25. Mathew C.C. The isolation of high molecular weight eucariotic DNA// methods in molecular biology / ED. Walker J.M. – N.Y.: Haman press. – 1984. – P. 31–34.
26. Nolan T, Hands RE, Bustin SA (2006). «Quantification of mRNA using real-time RT-PCR.». Nat. Protoc. 1: 1559–1582. DOI:10.1038/nprot.2006.236. PMID 17406449.
27. VanGuilder HD, Vrana KE, Freeman WM (2008). «Twenty-five years of quantitative PCR for gene expression analysis». Biotechniques 44: 619–626. DOI:10.2144/000112776. PMID 18474036.
ПРИЛОЖЕНИЕ
Приложение 1.
Приготовление рективов для выделения ДНК
Похожая информация.
Биологические микрочипы (biochips) или, как их чаще называют -microarrays, - это один из новейших инструментов биологии и медицины 21 века.
Введение
Изобретены биочипы были в конце 90-х годов в России и в США. Технология микрочипов – это принципиально новый уровень лабораторных исследований, так как она позволяет проводить одновременное тестирование тысяч образцов. Тысячи молекул ДНК или белков помещаются на стеклянные пластинки для создания ДНК- и белковых чипов соответственно. На основании экспериментальных данных сотрудниками лаборатории клеточной инженерии под руководством д.б.н. Белецкого И. П. Института Теоретической и Экспериментальной Биофизики (г. Пущино Московская обл.) была разработана технология для крупномасштабного изготовления стеклянных подложек при производстве ДНК-чипов, а также их использования при гибридизационном анализе ДНК. В его основе лежит использование комплементарного связывания нуклеотидов. Биочипы используются для самых разных целей. В медицине биочипы помогают за считанные часы обнаруживать у больных лекарственно устойчивые формы туберкулеза и лейкоза, а так же некоторые виды раковых заболеваний. Биочипы являются незаменимым инструментом для биологов, которые могут сразу, за один эксперимент, увидеть влияние различных факторов (лекарств, белков, питания) на работу десятков тысяч генов.
Биохимические микрочипы
Биохимические микрочипы, технологии производства и внедрения которых активно развиваются в России и за рубежом, являются сильнейшими из существующих инструментов для выявления и идентификации биологических материалов. В основе применения микрочипов лежит принцип быстрого определения взаимодействий тех или иных лигандов со множеством различных зондов одновременно. Собственно биологические микрочипы представляют собой ту или иную твердую подложку, на которой нанесены или определенные фрагменты нуклеиновых кислот, или белки, или углеводы, или какие-либо иные молекулы-зонды, способные быть узнанными или проявлять биологическую активность. Количество различных зондов на подложке может достигать сотен тысяч, причем чипы каждого типа строго идентичны и при существующих технологиях могут быть реплицированы в сотнях тысяч и миллионах копий нанесенных на подложку.
К главным причинам широкого распространения биочиповых исследований относят высокую чувствительность, специфичность и воспроизводимость, простоту процедуры выполнения, возможность одновременного анализа множества параметров и относительно невысокую стоимость работ. Эти же причины заставляют рассматривать биочипы как перспективный инструмент в различных областях народного хозяйства. Биочипы применяются для обнаружения бактериальных и вирусных контаминаций в продуктах питания, косметике и окружающей среде, выявления генно-модифицированных организмов в пищевых продуктах, диагностики и прогнозирования различных заболеваний, детекции особо опасных инфекционных агентов в анти-биотеррористических целях и др.
ДНК-микрочипы
ДНК–чипы представляют собой уникальный аналитический инструмент, позволяющий определять наличие в анализируемом образце (как правило, биологического происхождения) заданных последовательностей ДНК (т.н. гибридизационный анализ). Проведение анализа с помощью ДНК–чипов обходится в несколько раз дешевле, чем при использовании альтернативных технологий (электрофорез, ПЦР в реальном времени) и допускает, при наличии детектора несложной конструкции, работу вне лаборатории.
Впервые ДНК–чипы были использованы в исследованиях в конце 80-х годов прошлого века. В основе этого теперь уже широко распространенного метода, позволяющего одновременно анализировать экспрессию множества генов, лежит принцип узнавания мРНК-овых или кДНК-овых мишеней посредством их гибридизации с иммобилизованными на микрочипе одноцепочечными фрагментами ДНК.
ДНК–чип представляет собой твердую подложку, на которой иммобилизованы (как правило, ковалентно) однонитевые фрагменты ДНК разной длины: короткие – 15-25 нуклеотидов, длинные – 25-60 нуклеотидов и кДНК фрагменты – от 100 до 3000 нуклеотидов. В качестве материала подложки используют стекло, кремний, различные полимеры, гидрогели (например, на основе полиакриламида) и даже золото. Наиболее распространенные подложки – из стекла.
Белковые и пептидные чипы
Для анализа продуктов трансляции генов используют чипы, построенных на основе полипептидов. Большинство лекарственных мишеней являются белками, следовательно, белковые и пептидные чипы могут быть полезны для поиска новых лекарств. Белковые микрочипы могут оказаться чрезвычайно полезными в медицине в качестве миниатюрных аналитических систем для определения иммунного статуса организма, выявления аллергической сенсибилизации и идентификации специфических аллергенов. Микрочипы, представляющие собрание основных антигенов главных патогенных организмов (бактерии, грибы и вирусы), позволяют анализировать образцы крови на присутствие одновременно сотен, тысяч антител и быстро идентифицировать инфекции.Большое значение в развитии белковых микрочипов имеют способы регистрации сигналов. К ним относятся: самый первый из известных методов – РИА (радиоиммунологический анализ), применяющий радиоактивную метку, иммуноанализ с использованием флуоресцентных меток – ФИА и иммуноферментный анализ (ИФА), в котором меткой является молекула фермента, ковалентно связанная с молекулой антитела. В качестве меток в ИФА выбираются высокоактивные стабильные ферменты (щелочная фосфатаза, пероксидаза и др.). Преимуществом ИФА является возможность многократного усиления сигнала. В последние годы разработаны чувствительные системы субстратов, дающих нерастворимые флуоресцирующие продукты, например, ELF-97. Очевидно, что процесс изготовления белкового микрочипа должен включать процедуру закрепления, иммобилизации на микрочипе. Выбор метода определяется многими параметрами - природой исходного субстрата, последующей областью применения микрочипа и т.д. Белковые микрочипы активно применяются, прежде всего, для анализа всех известных (и доступных) биологических жидкостей, включая сыворотку/плазму крови, мочу, цереброспинальную жидкость, слюну, слезную жидкость, амниотическую жидкость, и др.
Углеводные микрочипы
Многие природные биомолекулы (белки, липиды) модифицированы сахарными остатками. Часто биологические процессы включают связывание сахаров с рецепторами, и микрочипы могут стать важным инструментом в исследовании таких взаимодействий. Гликолипиды, нанесенные на нитроцеллюлозу или поливинилиденфторид, представляют пример углеводных микрочипов. Эти гликолипиды взаимодействуют с белками с известной углевод-связывающей специфичностью для подтверждения предсказанных олигосахарид-белковых взаимодействий. Связывание углеводов с мембранами регистрируют с помощью флуоресцентно меченых гликолипидов. Углеводный состав связавшегося компонента определяют in situ масс-спектрометрически. Важно отметить, что олигосахариды, связанные с липидом, могут иметь различное происхождение, например, гликопротеины, протеогликаны, гликолипиды, целые клетки и синтетические олигосахариды. Таким образом, по взаимодействию олигосахаридов, последовательность которых известна, с мембраной могут быть идентифицированы конкретные связанные с сахарами белковые мотивы. И, наоборот, путем связывания неизвестных олигосахаридов с мембраной можно отобрать белки с известной структурой, чтобы определить, с какими олигосахаридами они связаны.
Тканевые микрочипы
Данные по анализу экспрессии генов только начинают давать нам важную информацию о биологической функции генов, их потенциальном клиническом влиянии или их пригодности в качестве мишени для лекарства. В то же время традиционный гистологический анализ образцов ткани требует больших затрат времени: ткани выдерживают в формалине, помещают в парафин, делают срезы и только затем красят и проводят микроскопический анализ на индивидуальных стеклах. В 1998 году для такого анализа впервые были изготовлены тканевые микрочипы посредством нанесения на одну подложку многих образцов ткани.
Изготовление микрочипа включает объединение до тысячи иголочных биопсий, взятых из помещенных в парафин образцов ткани, в парафиновом блоке с определенными координатами. Из этого блока делается до 300 срезов, которые переносятся на стекло для прокрашивания и анализа. Таким образом, из одного блока может быть произведено до 300 тысяч анализов. При этом такой способ вызывает минимальное повреждение ткани.
Приготовленные один раз, микрочипы могут быть испытаны на взаимодействие с различными молекулярными мишенями – ДНК, РНК или белками – в сотнях или даже тысячах образцах ткани. Принципиальное отличие тканевых чипов от, например, ДНК-чипов заключается в том, что в последнем случае определяется экспрессия тысяч генов в одной ткани/образце, тогда как в первом – один ген в тысяче различных тканей/образцов. Преимущество анализа с использованием тканевых микрочипов состоит в том, что все образцы ткани обрабатываются одинаковым способом, т.е. концентрации реагентов, время инкубации, температура и состав растворов неизменны. При этом для анализа требуется всего десятые или сотые доли миллилитра реагентов.
Тканевые микрочипы довольно активно используются для поиска маркеров, ассоциированных с теми или иными заболеваниями, в первую очередь, с онкологическими. Показаны примеры успешного применения тканевых чипов для анализа аутоиммунных заболеваний, сердечной недостаточности, диабета и нейродегенеративных патологий.
Порядок проведения анализа ДНК с использованием биочипов
Олигонуклеотидный зонд, модифицированный фосфатной группой по 5-концу, пришивали к стеклу с помощью конденсирующего агента (карбодиимида в присутствии имидазола). Затем проводили гибридизацию с олигонуклеотидным фрагментом, меченным биотином, и цветную пероксидазную реакцию с помощью стрептавидин-пероксидазного конъюгата. По интенсивности окраски окисленного субстрата – диметиламинобензидина (ДАБ) – судили о результате анализа.
Изготовление биологических чипов
На основании экспериментальных данных была разработана технология для крупномасштабного изготовления стеклянных подложек при производстве ДНК-чипов. Для широкого применения чиповых технологий необходимы простые, дешевые и эффективные методы изготовления в больших масштабах и контроля модифицированных подложек, содержащих поверхностные аминогруппы.. Наиболее часто используемыми твердыми подложками для изготовления микрочипов являются стекла, обработанные различными аминоалкилтриалкоксисиланами, например APTES (3-аминопропилтриэтоксисилан, Рис.3) для получения модифицированной поверхности.
Рис.3 Структурная формула APTES. а-в свободном состоянии, б-модифицированная на стекле
Такая поверхность является гидрофобной и позволяет наносить зонды с максимальной плотностью. Как следует из литературных данных, условия получения амино-модифицированной стеклянной подложки могут быть весьма разнообразными. Это касается как концентрации APTES, так и выбора растворителя, который, в свою очередь, может содержать различное количество воды. Для изготовления амино-модифицированных подложек в больших масштабах желательно использовать как можно менее токсичные и дешевые растворители, такие как ацетон и этанол. Показано, что природа органического растворителя практически не влияет на качество получаемых стекол. Активация нуклеиновых кислот осуществляется при помощи карбодиимидов(CDI)- это один из ранних методов получения аффинных сорбентов, который до сих пор остается достаточно распространенным. В основе метода лежит способность карбодиимидов активировать концевую фосфатную группу нуклеиновых кислот. Активированные карбодиимидом олигонуклеотиды легко вступают в реакцию с амино- или сульфгидрильными группами носителей. К настоящему времени достаточно подробно изучен механизм активации карбодиимидами фосфатных групп олигонуклеотидов. Лимитирующей стадией реакции является протонирование карбодиимидов с образованием промежуточных неустойчивых реакционноспособных О-фосфорилизомочевины. Достаточно сильные нуклеофильные группы носителя могут ингибировать эту стадию

Кроме того, протонированные нуклеофильные группы не способны вступать в реакцию с соединениями 1 [Рис.4]. Вследствии этого выход конечного продукта 2 при использовании полимеров с высокоосновными группами может уменьшиться. Замещенные О-фосфорилизомочевины 3 способны также трансформироваться в побочные продукты, например гидролизоваться с высвобождением исходной фосфатной группы и мочевины, либо претерпевать ряд последовательных превращений с образованием пирофосфатов, полифосфатов и производных О-фосфорилизомочевины. Последние, в свою очередь, могут дать нереакционноспособные производные N-пирофосфорилмочевины 3 или N-фосфорилмочевины 4.
Для увеличения эффективности иммобилизации нуклеотидов на полимер активацию карбодиимидами проводят в присутствии нуклеофильного катализатора – имидазола, образующий соответствующий фосфамид олигонуклеотидов, который после протонирования остатков азола является высокореакционноспособным соединением по отношению к нуклеофилам. Чаще всего получаются фосфоимидазолиды 5 (Рис. 5)

Преимуществом этого подхода является отсутствие ряда побочных реакций, поскольку производное 5 стабильнее, чем О-фосфорилмочевина 1. Соединение 5 более эффективно взаимодействует с аминогруппой носителя, что способствует увеличению выхода при иммобилизации.
Проведение гибридизации.
В основе гибридизационного анализа лежит использование комплементарного связывания нуклеотидов (Рис.6,б). Известные в настоящее время реакции можно разделить на две большие группы. Первая группа методов основана на амплификации – циклически повторяющейся репликации in vitro искомого фрагмента ДНК (РНК). Амплифицированный фрагмент затем выявляется путем электрофореза в геле. Ко второй группе относятся методы прямого обнаружения специфической последовательности нуклеотидов (ДНК или РНК) при помощи коротких олигонуклеотидных одноцепочечных фрагментов, имеющих репортерную группу (зонд). Эти реакции получили название ДНК-зондирования. Их чувствительность зависит от вида используемого зонда и может быть весьма высокой. Наиболее распространенной нерадиоактивной репортерной группой, включаемой в состав олиго- и полинуклеотидных зондов, является витамин биотин (природный кофактор группы ферментов – карбоксилаз) (Рис 6,А)

Рис. 6 Схема метода гибридизационного анализа на примере идентификации генетически модифицированных источников растительного происхождения. А – ПЦР с использованием праймеров меченных биотином - индекс (мБ); Б – гибридизация ПЦР продуктов со специфическими олигонуклеотидами, иммобилизованными на биологическом микрочипе - индекс (и)
Биотинилированное производное дУТФ (био-УТФ) включается в образуемые ДНК-полимеразами цепи вместо тимина и способно к комплементарным взаимодействиям с аденином.

Биотин – соединение, стойкое к действию высоких температур, к кислой и щелочной среде, хорошо растворяется в воде и спирте. Он является коферментом во многих реакциях присоединения (карбоксилирования). Биотин легко может вступать в стойкое соединение с различными белками, в том числе с ферментами и иммуноглобулинами. В большом количестве биотин содержится в белках птичьих яиц, где он связан с гликопротеидом авидином, имеющим молекулярную массу 68 кДа. Биотин обладает чрезвычайно высоким сродством к авидину, а также к его бактериальному аналогу – стрептавидину (константа диссоциации комплекса биотин-авидин 10-15 М) и образуeт чрезвычайно стойкий комплекс (КА=10 в 15-той степени на моль в минус 1-й степени).
Проведение пероксидазной реакции
Для проведения пероксидазной реакции используется коньюгат, состоящий из фермента, связанного со стрептавидином. Этот коньюгат образует очень прочный комплекс биотин-стрептавидин, который используется как связующий мостик между образовавшимся в ходе гибридизации дуплексом ДНК и ферментной меткой.
Наибольшее распространение в качестве ферментной метки получила пероксидаза хрена, которую впервые применили Накане и Пирс. Одна молекула фермента, конъюгированная со стрептавидином, способна "обработать" большое количество молекул субстрата. Под действием пероксидазы, субстрат H2O2 образует с красителем диаминобензидином (ДАБ) нерастворимый в воде и спирте комплекс коричневого цвета, который накапливается вокруг фиксированного дуплекса.
Обработка результатов анализа
Визуализацию гибридизационной картины на биологическом микрочипе для анализируемой пробы осуществляют с помощью аппаратно-программного комплекса для анализа биологических микрочипов "EuroBioWto Биоконтроль" и компьютерной программы "MedGen". Полученную на экране компьютера гибридизационную картину для анализируемой пробы сравнивают с гибридизационной картиной для положительного контроля (заведомо трансгенной ДНК) и гибридизационной картиной для отрицательного контроля (заведомо нетрансгенной ДНК). Пример схемы гибридизационной картины биологического микрочипа приведен на рис.9.

Рис. 9 Пример схемы биологического микрочипа для идентификации генетически модифицированных источников растительного происхождяения.PC положительный контроль, NC отрицательный контроль, Lect-ген белка сои, Zein-ген белка кукурузы, Gus, Nos, 35s, Npt, Ocs-трансгенные последовательности
Наличие яркого специфического окрашивания биологического микрочипа в местах, содержащих иммобилизационные олигонуклеотиды (для промоторов 35S и Nos, терминатора Ocs, генов Gus или NptII) , свидетельствует о присутствии конкретных чужеродных последовательностей ДНК в анализируемой пробе, т. е. о трансгенности анализируемой ДНК. Отсутствие окраски после анализа указывает на отсутствие конкретных чужеродных последовательностей ДНК в анализируемой пробе, т. е. о нетрансгенности анализируемой ДНК. Наличие окрашивания NC cвидетельствует о получении ложноположительного результата. Причиной может быть загрязнение ГМИ реактивов и/или оборудования. В этом случае необходимо обработать поверхности лабораторных столов и оборудования раствором соляной кислоты (1 моль/дм в кубе), заменить реактивы на свежеприготовленные и повторить анализ.
Анализ с использованием биочипов находит в настоящее время широкое применение на практике. Он отличается высокой чувствительностью, простотой процедуры выполнения, возможностью одновременного анализа множества параметров, специфичностью и воспроизводимостью. Применение биологических микрочипов может быть очень эффективно в таких сферах деятельности, как: обнаружение контаминации в продуктах питания и окружающей среде, диагностика и прогнозирование заболеваний, обнаружение элементов биологического оружия, оценка лекарственной эффективности. Разработка биочиповых технологий в РФ является одной из тех высоко-технологичных областей, которой уделяется особое внимание.
Список литературы
1. Шляпникова Е. А. , Шляпников Ю. М. , Афанасьев В. Н. , Афанасьева Г. В. , Гаврюшкин А. В. , Белецкий И. П. . Исследование качества амино-модифицированных подложек, используемых для гибридизационного анализа.// Биоорганическая химия 2006 №4 С.68-86.
2. Шляпникова Е. А., Шляпников Ю. М., Грановский И. Э., Афанасьева Г. В., Гаврюшкин А. В., Белецкий И. П. "Биологические микрочипы в медицине" Институт теоретической и экспериментальной биофизики РАН.
3. Timofeev E., Mirzabekov A. (1998) Immobilization of DNA in polyacrylamide gel for the manifacture of DNA and DNA-oligonucleotide microchips and protein microchips. Anal.Biochem., 259, 34-41.
4. Шишкина И. Г., Левина А. С., Зарытова В. Ф. "Аффинные сорбенты, содержащие нуклеиновые кислоты и их фрагменты" //Успехи химии// 2001. №70(6).С.581,.
5. Makretsov N.A., Huntsman D.G., Nielsen T.O., Yorida E., Peacock M., Cheang M.C., Dunn S.E., Hayes M., van de Rijn M., Bajdik C., Gilks C.B. (2004) Hierarchical clustering analysis of tissue microarray immunostaining data identifies prognostically significant groups of breast carcinoma. Clin Cancer Res., 10(18 Pt 1), 6143-6151.
6. Hu S., Loo J.A., Wong D.T. (2006) Human body fluid proteome analysis. Proteomics, 6(23), 6326-6353.
7. Ekins R.P. (1987) An overview of present and future ultrasensitive nonisotopic immunoassay development. Clin. Biochem. Revs. 8, 12-22.
8. Fukui S. et al. (2002) Oligosaccharide microarrays for high-throughput detection and specificity assignments of carbohydrate-protein interactions. Nat.Biotechnol.20, 1011-1017.
9. Kononen J. et al. (1998) Tissue microarrays for high-throughput molecular profiling of tumour specimens. Nat.Med. 4, 844-847/
10. Wang et al. (2002) Carbohydrate microarrays for the recognition of cross-reactive molecular markers of microprobes and host cells. Nat.Biotechnol.20, 275-281.
11. Perkel J.M.(2002) Tissue microarrays: advancing clinical genomics. The Scientist 21, 39.
12. Helmchen B., Weckauf H., Ehemann V., Wittmann I., Meyer-Scholten C., Berger I. (2005) Expression pattern of cell cycle-related gene products in synovial stroma and synovial lining in active and quiescent stages of rheumatoid arthritis. Histol Histopathol., 20(2):365-372.
13. Hueber W., Kidd B.A., Tomooka B.H., Lee B.J., Bruce B., Fries J.F., Sonderstrup G., Monach P., Drijfhout J.W., van Venrooij W.J., Utz P.J., Genovese M.C., Robinson W.H. (2005) Antigen microarray profiling of autoantibodies in rheumatoid arthritis. Arthritis Rheum.; 52(9), 2645-2655.
14. Mobasheri A., Airley R., Foster C.S., Schulze-Tanzil G., Shakibaei M. (2004) Post-genomic applications of tissue microarrays: basic research, prognostic oncology, clinical genomics and drug discovery. Histol Histopathol., 19(1), 325-335.
15. Мелентьева Г.А.. Фармацевтическая химия. Том 2, Москва "Медицина" 1976. С.68
Гибридизационный анализ ДНК с использованием биологических микрочипов